โครงการ CASAVASTORE
วัตถุประสงค์คือเพื่อศึกษาข้อมูลฟีโนไทป์ จีโนไทป์ และ สรีรวิทยาเกี่ยวกับการพัฒนาของรากสะสมอาหารของมันสำปะหลังเพื่อนำไปสู่การพัฒนาเทคโนโลยีเกษตรแม่นยำ โดยมีแผนงานวิจัย 4 แผนงาน ดังนี้
หน่วยงานความร่วมมือ : ไบโอเทค สวทช., เนคเทค สวทช., มจธ. , ศูนย์วิจัยพืชไร่ระยอง (ศวร.)
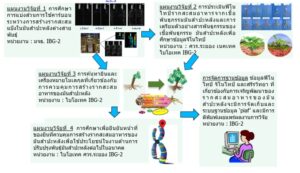
แผนงานวิจัยที่ 1 การศึกษาการแบ่งส่วนการใช้คาร์บอนระหว่างการสร้างรากสะสมแป้งในมันสำปะหลังต่างสายพันธุ์ หน่วยงานที่รับผิดชอบคือ มจธ. จูลิช-IBG2
แผนงานวิจัยที่ 2 การประเมินฟีโนไทป์รากสะสมอาหารจากเชื้อพันธุกรรมมันสำปะหลังและการเตรียมตัวอย่างสารพันธุกรรมของเชื้อพันธุกรรม มันสำปะหลังเพื่อศึกษาข้อมูลจีโนไทป์ หน่วยงานที่รับผิดชอบคือ ศวร.ระยอง เนคเทค ไบโอเทค และจูลิช-IBG2
แผนงานวิจัยที่ 3 การค้นหายีนและเครื่องหมายโมเลกุลที่เกี่ยวข้องกับการควบคุมการสร้างรากสะสมอาหารของมันสำปะหลัง หน่วยงานที่รับผิดชอบ คือ ไบโอเทค และ จูลิช-IBG 2
แผนงานวิจัยที่ 4 การศึกษาเพื่อยืนยันหน้าที่ของยีนที่ควบคุมการสร้างรากสะสมอาหารของมันสำปะหลังเพื่อใช้ประโยชน์ในงานด้านการปรับปรุงพันธุ์มันสำปะหลังต่อไปในอนาคต หน่วยงานที่รับผิดชอบคือ ไบโอเทค ศวร.ระยอง และจูลิช-IBG2
การจัดการฐานข้อมูล ข้อมูลฟีโนไทป์ จีโนไทป์ และสรีรวิทยา ที่เกี่ยวข้องกับการเจริญพัฒนาของรากสะสมอาหารของมันสำปะหลังจะมีการจัดเก็บและระบบฐานข้อมูล ‘piaf’ และมีการตีพิมพ์เผยแพร่ผลงานการวิจัย หน่วยงานที่รับผิดชอบคือ จูลิช-IBG-2 และสวทช.
ความก้าวหน้า
ปัจจุบันได้ปลูกมันสำปะหลัง จำนวน 600 สายพันธุ์ (crop ละ 100 สายพันธุ์) แล้ว ได้มีการใช้ซอฟต์แวร์วัดประเภทราก (รากฝอย รากที่เริ่มโป่งพอง รากสะสมอาหารระยะเริ่มต้น และ รากสะสมอาหาร) เพื่อวิเคราะห์หาว่าพันธุ์ไหนมีรากสะสมอาหารเกิดได้เร็ว ปานกลาง หรือช้า ใน 600 สายพันธุ์ และคัดเลือกพันธุ์ที่สนใจจำนวน 100 สายพันธุ์ มาปลูกเปรียบเทียบในแปลงเดียวกันเพื่อเป็นการยืนยัน ได้ทำการเลือก 100 สายพันธุ์ที่สนใจเพื่อนำมาปลูกทดสอบใน crop 7
สวทช. ร่วมกับมหาวิทยาลัย RWTH Aachen สหพันธ์สาธารณรัฐเยอรมนี ได้พัฒนากรรมวิธีการสกัดดีเอ็นเอสำหรับมันสำปะหลังให้ได้คุณภาพสูง เพื่อใช้เทคนิค De novo Oxford Nanopore sequencing ในการวิเคราะห์ลำดับเบส ซึ่งจะได้ข้อมูลขนาดจีโนมระดับใหญ่และละเอียดมากยิ่งขึ้นได้สกัดดีเอ็นเอจากใบอ่อนมันสำปะหลัง จำนวน 592 สายพันธุ์ และส่งตัวอย่างดีเอ็นเอให้ IBG-2 จำนวน 409 สายพันธุ์ และอยู่ระหว่างส่งตัวอย่างอีก 183 สายพันธุ์ IBG-2 ได้วิเคราะห์ลำดับเบสแล้วจำนวน 260 สายพันธุ์ พบว่า มันสำปะหลังมีขนาดจีโนมประมาณ 740-760 Base pair (bp) ยังไม่สามารถวิเคราะห์ GWAS ได้ เนื่องจากข้อมูลการวิเคราะห์ลำดับเบสยังไม่ครบ 600 สายพันธุ์ จึงยังไม่สามารถหาความสัมพันธ์ระหว่าง genotype กับ phenotype ได้